摘要
生物多样性丧失与保护是当今全球最受关注的议题之一,目前普遍认为由于人类活动的影响导致了第六次生物大灭绝的到来。而我国野生动物保护形势更为严峻,其中,濒危旗舰动物保护是野生动物保护的主要抓手,对生物多样性保护和生态系统平衡具有重要意义。本次沙龙邀请到中国科学院动物研究所胡义波研究员就“濒危旗舰动物研究与保护”开展主旨报告。主旨报告从脊椎动物受威胁现状、旗舰物种定义及其保护意义、代表性濒危旗舰动物研究与保护、濒危动物保护存在的问题等方面,系统地介绍了我国近年来在濒危动物保护生物学领域取得的一系列重要成果,以及面临的一些急需解决的问题及建议解决方案。
关键词:濒危动物;旗舰动物;种群;栖息地;遗传多样性
濒危动物保护的历史可以追溯到20世纪中叶,当时随着工业化和城市化的快速发展,人类活动对自然环境的影响日益加剧,导致许多物种面临灭绝的威胁。为了应对这一挑战,国际社会开始采取行动。1948年,国际自然保护联盟(International Union for Protection of Nature, IUPN, 后来更名为 International Union for Conservation of Nature, IUCN)成立,开始发布濒危物种红皮书,以唤起全球对野生物种生存现状的关注,并为各国政府提供数据参考。我国也在1988年颁布了《中华人民共和国野生动物保护法》,并在随后的年份中进行了多次修订,以加强对濒危野生动物的保护。在我国,自上世纪50年代建立第一个自然保护区开始,到如今已逐步建立起了包括国家公园、自然保护区、自然公园等在内的自然保护地体系,各类自然保护地总数达到1.18万处,陆域自然保护地总面积约占陆地国土面积的18%以上。这为保护生物多样性、维护生态平衡发挥了重要的作用。同时野生动物保护离不开科技支撑,对濒危物种尤其是濒危旗舰动物的研究与保护能为政府部门制定更加科学的保护策略提供有价值的参考。
一、脊椎动物受威胁现状
由于人类活动的影响,许多科学家和研究者都认为我们已经进入第六次生物大灭绝时期1,2。自1500年以来,地球可能已经失去了200万种已知物种的7.5%至13%,即惊人的15万至26万种物种,《全球生物多样性和生态系统服务评估报告(2019)》指出其中至少有680种脊椎动物灭绝3。
我国脊椎动物受威胁状况高于全球水平。据《中国生物多样性红色名录——脊椎动物卷(2020)》评估显示,37.5%的两栖动物、30.8%爬行动物、25.9%哺乳动物、22.3%的鱼类及11.2%的鸟类处于受威胁状态,总计22.02%的脊椎动物处于受威胁等级,而全球受威胁的脊椎动物比例为20%。我国受威胁的脊椎动物共有1050种,其中562种为易危等级(Vulnerable,VU),290种为濒危等级(Endangered,EN),198种为极度濒危等级(Critically Endangered,CR)。在2021年发布的《国家重点保护野生动物名录》中,脊椎动物占比为86.8%。其中哺乳纲包含185种,鸟纲包含394种,两栖纲包含93种,爬行纲包含94种,鱼纲包含92种。
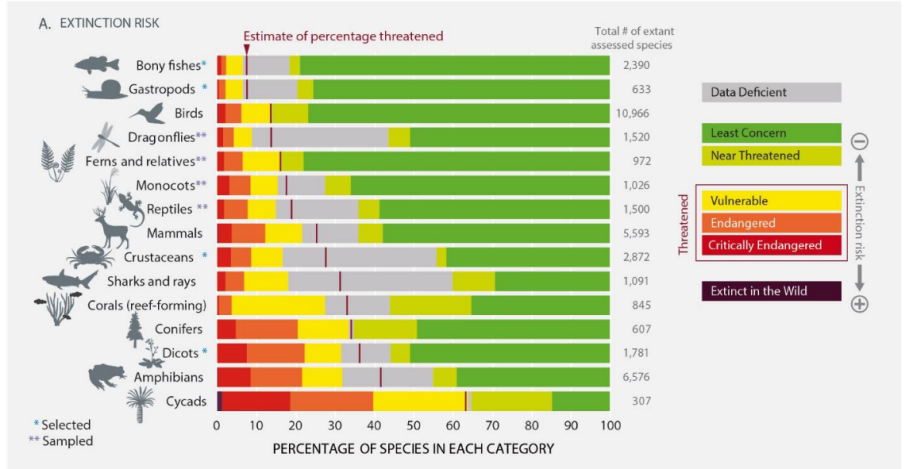
图1全球不同物种类群的灭绝风险
来源:(Díaz et al. 2019, IPBES)
二、旗舰物种的保护现状及意义
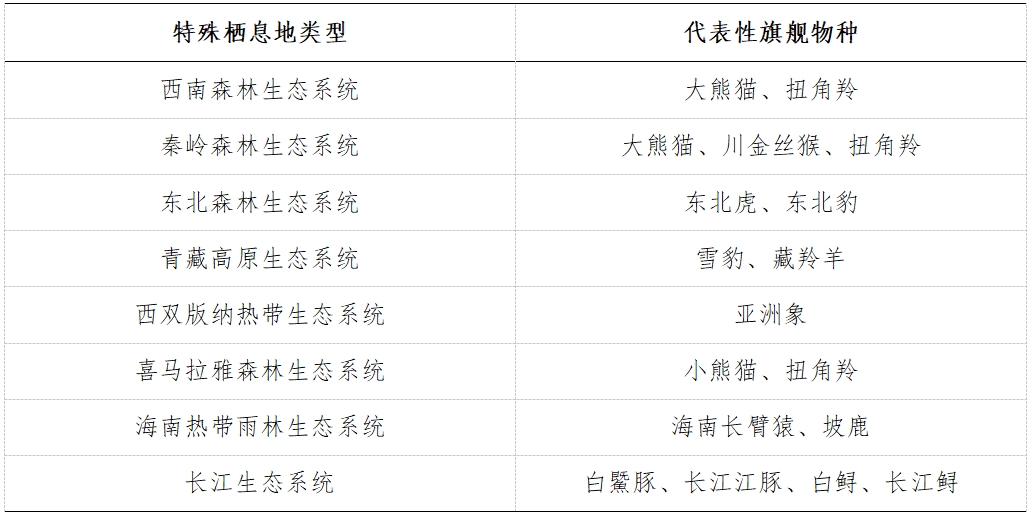
关于旗舰物种(Flagship species)的定义通常有两种:1)是一类利用自身“魅力”来获取大众关注和支持的物种,该物种对社会生态保护力量具有特殊号召力和吸引力,可促进社会对物种保护的关注4,例如大熊猫、小熊猫、朱鹮、麋鹿和川金丝猴。对旗舰物种保护可以起到广告宣传作用,唤起公众对野生动植物及生物多样性保护的热情和关注,还能快速募集保护资金,支持野生动植物及生物多样性保护工作。2)指被挑选出来作为代表一个需要保护的特殊栖息地的代表物种。例如代表东北森林生态系统的东北虎和东北豹,代表青藏高原生态系统的雪豹和藏羚羊等物种(表1)。旗舰物种通常可以发挥伞护种(Umbrella species)的作用,既通过对旗舰物种的保护能够促进对该物种同域分布的其他众多物种的保护,在大熊猫“伞护效应”下,金丝猴、红豆杉等8000多种伴生珍稀动植物得到良好保护。
表1 特殊栖息地类型及其代表性旗舰物种
由旗舰物种保护带动建设的国家公园则为野生动物保护提供了更高水平的保护和管理,这有助于实现野生动物种群数量的稳定和恢复。第一批正式建设的五个国家公园保护面积达23万平方公里,涵盖近30%的陆域国家重点保护野生动植物种类(图2)。其中四个是由旗舰动物保护带动建设的,包括大熊猫、东北虎豹、海南热带雨林和三江源国家公园,其中大熊猫国家公园保护了70%以上的野生大熊猫,连通了13个破碎的大熊猫种群栖息地,这是分散的自然保护区所不具备的优势。

图2 首批国家公园分布示意图
来源:(星球研究所:https://mp.weixin.qq.com/s/4Qfr0VNdFTxWrRNFBQjoAw)
随着我国各种保护措施的实施,加上国家公园的建设,近年来我国旗舰物种的保护取得了显著的成绩,旗舰物种包括其他野生动物的数量持续增长。根据2022年发布的数据,我国野生大熊猫的数量从20世纪七八十年代的1114只增至1864只,三江源国家公园内的藏羚羊增长至7万多头、雪豹恢复到1200多只;东北虎、东北豹数量分别从试点之初的27只、42只增长到70只、80只左右,海南长臂猿野外种群数量从40年前的仅存2群不到10只增长到7群42只。东/西黑冠长臂猿的种群数量从不足百只增加至约700只;亚洲象的种群数量由1985年的180头增至300头左右;野生绿孔雀增至约550只;滇金丝猴种群数量增至3800余只。
三、代表性濒危旗舰动物研究与保护
在当前保护生物学的研究中,物种保护策略通常涵盖三个方面:种群保护、栖息地保护以及遗传多样性保护。越来越多研究显示,遗传多样性的丧失与近交会加速物种濒危灭绝。与未受威胁的物种相比,受威胁物种展现出显著较低的遗传多样性,这进一步强调了遗传多样性保护的重要性。“昆明—蒙特利尔全球生物多样性框架”的发布则是将野生动植物的遗传多样性保护首次纳入保护目标体系。然而,我国当前仅对9.3%的陆生脊椎动物物种进行了种群水平的遗传多样性评估,主要集中在哺乳类和两栖类,相比之下,鸟类和爬行类的评估工作较为薄弱,特别是鸟类,其遗传多样性评估率仅为4.08%。这表明,在遗传多样性保护方面,我国仍有大量工作有待开展。
1. 大熊猫的研究与保护
在众多脊椎动物中,大熊猫是一个典型的保护生物学研究模型。然而,从上世纪七八十年代以来直到现在,偶尔有一些悲观的论调认为,由于大熊猫种群数量少、繁殖能力低下、遗传多样性低和专食竹子等生物学特征,最终会致使其走向灭绝。事实是这样的吗?
全基因组和种群基因组数据分析结果表明大熊猫濒危主要是古气候变化和人类活动等外因所致 (图3)5。关于大熊猫种群数量的悲观情绪主要缘于上世纪80年代第二次大熊猫调查的结果,当时记录的数量为1114只,仅有首次调查数目的一半,从而引发了广泛担忧。但是随后实施的天然林禁止采伐、退耕还林还草等保护措施,则有效促进了其种群数量的恢复。第四次大熊猫调查结果显示其种群数量已增至1864只,这表明随着栖息地保护和恢复工作的推进,大熊猫种群数量呈增长趋势。此外,传统的调查方式低估了野外大熊猫的数量。传统调查大熊猫数量的方式为竹子咬节法结合地理距离法。如今,最新的分子粪便学技术为大熊猫种群数量调查提供了新手段。该技术利用大熊猫粪便表层的黏液细胞提取大熊猫DNA,从而实现个体识别。通过此方法,对四川王朗保护区的大熊猫调查显示,该区域有66只个体,但是第三次全国大熊猫调查时仅为27只,这表明野生大熊猫的实际种群数量高于之前估计值6。
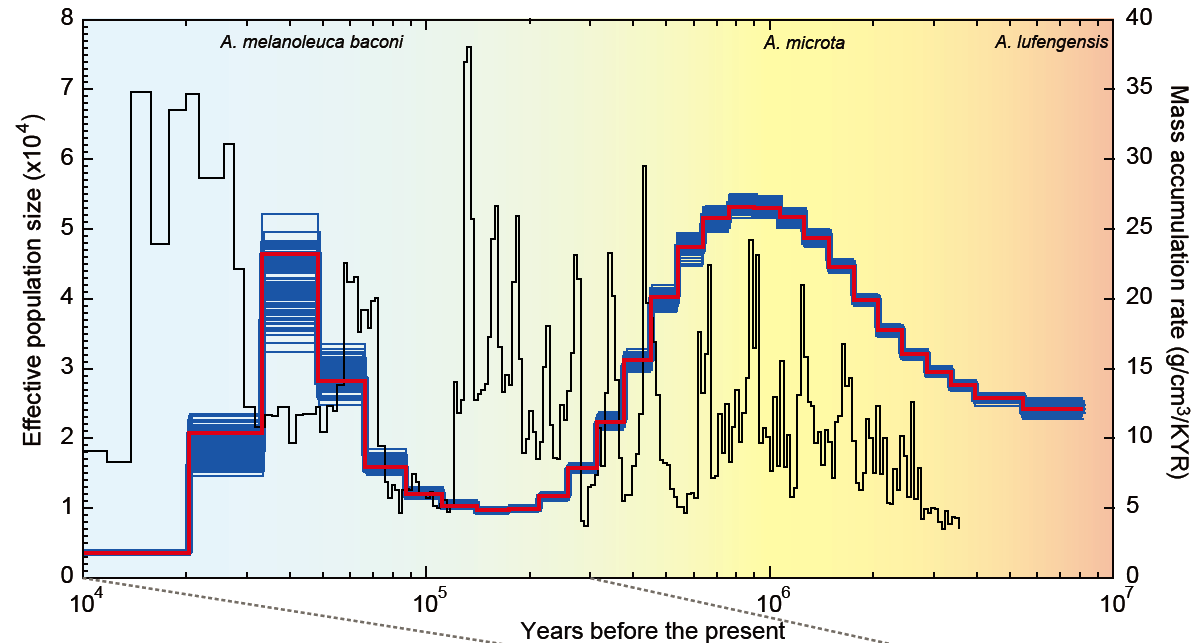
图3 大熊猫种群历史动态变化
来源: (Zhao et al. 2013, Nature Genetics)
在大熊猫繁殖能力方面,过去人们普遍认为大熊猫存在发情难、受孕难及幼崽存活率低的问题,但这是繁育技术不成熟产生的假象。胡锦矗先生在四川卧龙自然保护区长期开展野生大熊猫研究,对大熊猫求偶交配和繁殖育幼进行了详细观察,他发现野外大熊猫采用一雌多雄的竞争求偶交配模式,且求偶交配行为正常7。在发情期间,多只雄性大熊猫会进行竞争从而获得与雌性大熊猫交配的机会,有时会通过打斗来决定胜负,这可能会在大熊猫的脸上留下疤痕,有意思的是这也成为现在使用红外相机识别大熊猫个体的特征之一。另外使用红外摄像机观测野外大熊猫产仔育幼行为,发现野外大熊猫能够进行正常的繁育行为。2001年,潘文石先生等人在陕西长青自然保护区长期开展野生大熊猫生态学研究时,发现一个英雄大熊猫妈妈,成功繁育了4个后代。在大熊猫圈养繁育工作中,目前已经解决了大熊猫圈养繁殖三难的问题,并取得了巨大成功。圈养大熊猫种群数量增长迅速,从2013年的375只到2024年1月的728只,目前圈养繁育策略已经从重数量向重质量转变。在遗传多样性方面,基于微卫星标记和全基因组SNP标记的研究结果表明,与其它濒危物种相比,大熊猫仍具有较高的遗传多样性和演化潜力,大熊猫遗传多样性并不低8,9。
大熊猫食性特化、专食竹子是对大熊猫命运保持悲观论调的另外一个原因。然而,研究表明大熊猫针对取食竹子已经演化出了一套特殊的形态和生理特征,如演化出了伪拇指用来抓握竹子(图4)10,并且大熊猫具有发达的颧弓和面部肌肉、臼齿,便于咀嚼竹子。在觅食对策上,大熊猫会优先选择营养最好的竹子种类和部位。同时,为了减少能量消耗,大熊猫的休息时间更长,巢域面积小,还避免在茂密的竹林和灌丛中穿行活动。研究表明,野生和圈养大熊猫平均每日能量消耗率为5.2 MJ/day, 仅为体型预测值的37.7%, 这比考拉(69%)低, 与树懒(36%)相似11。同时,大熊猫体外的厚毛皮也有助于保持恒定体温,这可以进一步减少能量消耗(图5)11。另外,大熊猫和肠道微生物的互作促进了竹子营养利用,如:大熊猫肠道微生物具有消化纤维素的细菌,及消化纤维素和半纤维素的酶基因12;竹子中黄酮类化合物可调控大熊猫肠道微生物组成和功能,降低致病菌丰度,有利于维持大熊猫肠道微生物健康稳态;肠道微生物可同步化大熊猫肝脏节律,赋予大熊猫脂类代谢可塑性,以补偿机体对脂类物质的需求等13。
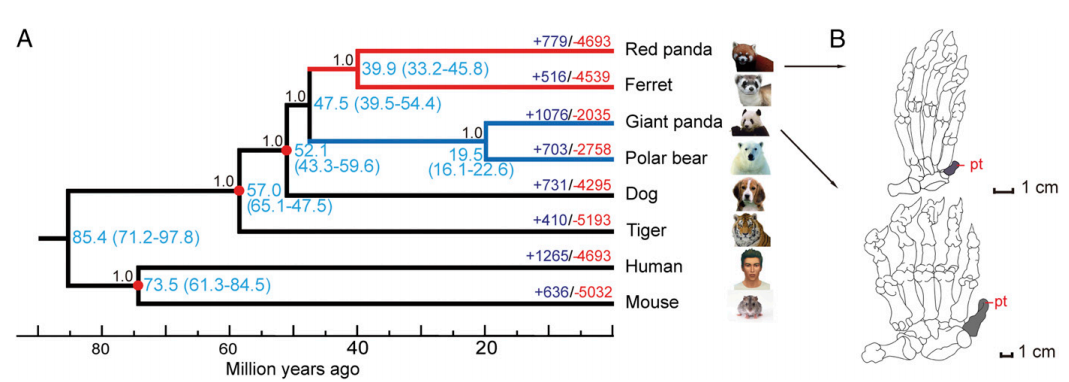
图4 大熊猫和小熊猫的系统发育树(A)及趋同进化的伪拇指(B)
来源:(Hu YB et al. 2017, PNAS)
图5 大熊猫厚毛皮使其保持恒定的体温
来源:(Nie YG et al. 2015, Science)
专食竹子对大熊猫最大的影响是,若竹子资源枯竭,大熊猫是否面会临灭绝风险?如上世纪七十年代,因大规模竹子开花,岷山和邛崃等山系都出现了野外大熊猫个体死亡数量增多的现象。那么历史上大规模竹子开花是否会对大熊猫种群造成严重的影响呢?通过对1988年前后野生大熊猫皮张及现存种群粪便中的遗传信息对比分析发现,现生大熊猫的遗传多样性并未显著降低、有效种群大小未显著下降,也未经历近期的种群瓶颈14。在野外,大熊猫面对大规模竹子开花时,会表现出两种应对策略:1)当一种竹子开花时,大熊猫会觅食其他种类的竹子;2)大熊猫可迁徙至其他区域寻找竹子。因此,大熊猫国家公园的建设将破碎的栖息地连接起来,便能很好地应对此类事件对大熊猫种群的影响。此外,考虑到竹子开花周期为60年左右,因此大熊猫种群遇到野外大规模竹子开花的概率相对较低。
结合多年对大熊猫的科学研究结果显示,野生大熊猫的繁殖行为正常且具有较高的繁殖率和遗传多样性,并且其野外种群数量正逐步增长,显示其仍具备演化潜力。2016年IUCN将大熊猫的濒危等级从“濒危”下调为“易危”,则是对我国多年来大熊猫保护成效的认可。然而,濒危等级降级并不意味着保护工作可以放松,仍需持续关注与加强保护。
2. 小熊猫的研究与保护
小熊猫作为另一旗舰物种,常与大熊猫产生误解,需明确小熊猫并非大熊猫的幼崽,而是独立物种。长期以来,小熊猫的属下分类地位存在争议,是分为两个亚种还是两个物种存在争议,分类地位的不清晰会导致保护对策的制定缺乏科学性。为解决这一争议问题,研究人员对小熊猫主要分布区进行了遗传样本的采样。结果发现,全基因组SNPs、母系遗传的线粒体基因组和父系遗传的Y染色体SNPs等三种遗传标记分析结果均支持小熊猫野生种群可分为两个物种,即喜马拉雅小熊猫和中华小熊猫 (图6), 这两个物种的分布并非以怒江为界,最可能是受到雅鲁藏布江地理障碍的影响15。随后,印度学者在我国藏南地区及印度锡金采集了小熊猫的新鲜粪便,并进行了线粒体分子标记的分析,构建了线粒体单倍型的系统发育树。其结果支持小熊猫两个物种的边界是雅鲁藏布江16。
通过对小熊猫保护基因组学的研究,科研人员认为应该科学地划分保护管理单元,中华小熊猫可至少分为藏东南-高黎贡、小相岭-凉山、邛崃等3个管理单元;喜马拉雅小熊猫的遗传多样性较低、近交和有害变异水平较高,是最需要迫切保护的物种;并且由于两个物种之间已经发生了显著的遗传分化,因此应避免圈养条件下的杂交,从而保存其独特的遗传多样性,有助于未来复壮野生小种群;此外,两种小熊猫分布涉及5个国家,为了有效保护小熊猫种群,非常有必要开展跨境保护合作。

图6 中华小熊猫(A, C)与喜马拉雅小熊猫(B, D)
来源:(Hu YB et al. 2020, Science Advances)
3. 虎的研究与保护
我国在虎的遗传学研究与野外生态学监测方面的工作尤为突出。研究表明,虎被分为多个亚种,基因组学上证据支持现生虎可分为六个亚种。值得注意的是,这些亚种的分化时间相对较晚,大约在最近2万年才开始逐渐分歧,其中苏门答腊虎是最早分化出的亚种17。华南虎在野外已难以寻觅,但尚未宣布功能性灭绝,其在动物园中仍有圈养种群。在野生东北虎豹的监测方面,我国也取得了显著成果。北师大团队在东北虎豹国家公园内架设了高清摄像头、无人机和红外相机等,实现了对东北虎豹的全域覆盖监测。这使得我们对东北虎豹的活动规律和个体数量有了较为清晰的了解。未来,综合运用现代有线无线融合数据传输网、卫星遥感与无人机、野生动物多样性与生态要素自动采集、人工智能识别、大数据挖掘与云计算等现代信息技术手段,有望构建天地空一体化的生态物联网监测体系。这将为野生动物的保护提供更有力的支持。
4. 扭角羚的研究与保护
扭角羚作为有蹄类物种中的旗舰物种,其分布范围广泛,从喜马拉雅山脉一直延伸至我国秦岭山系,但其属下分类问题一直存在争议。研究人员通过四种分子标记提供的证据,发现扭角羚可分为中华扭角羚和喜马拉雅扭角羚两个系统发生物种,其中,中华扭角羚又可以分为秦岭亚种和四川亚种,而喜马拉雅扭角羚则分为高黎贡亚种和不丹亚种18。这些物种和亚种在形态和毛色上均存在明显的区别,且种群遗传结构研究也识别了它们之间的地理分布边界。科学划分这些亚种和物种有助于制定更精准的保护策略,指导野化放归以及生态廊道的建设等。遗传多样性评估也显示,秦岭亚种是最需要迫切保护的亚种。
5. 长江江豚的研究与保护
长江江豚是目前长江生态系统中仅存的大型旗舰物种。然而其分类地位也是近年来才被确定下来。长江江豚曾经被认为是窄脊江豚中的长江亚种,2018年,种群基因组学研究发现,长江江豚在分子系统树中是一个单系群,显著不同于海洋生活的窄脊江豚东亚亚种,因此建议作为两个系统发生物种19,这从遗传上明确了长江江豚的分类地位,从而可以对其制定针对性的保护策略。目前野外种群及栖息地监测、声学通讯研究被用于指导长江江豚的物种保护工作中,这些努力为长江江豚的保护工作奠定了坚实基础。长江十年禁渔,将使其未来保护前景变得更为光明。
四、濒危动物保护存在的问题及应对之策
虽然我国在濒危动物的保护与研究中取得了一些成果,但是仍然有许多亟待解决的问题。如我国目前有988种(类)国家重点保护野生动物,大多数保护动物的种群、栖息地及其遗传多样性情况并不清楚,其致危机制尚不明晰,缺乏深入的科学研究和全面的了解;对于濒危物种的解危技术,目前仍然处于不成熟、不系统的状态,尚未形成一套科学有效的解危体系,同时,针对已经灭绝的动物的复活技术体系亟需探索和突破;保护生物学领域的基础研究成果虽然丰硕,但将其转化为实际的保护实践却仍需加强,如何将这些科研成果更好地应用于具体的物种保护工作中,是我们需要加强着力的方面;此外,随着人类活动的不断扩展,人与野生动物包括濒危动物之间的冲突也日益加剧,对于这一问题的研究同样需要加强,以寻找更加和谐的人与野生动物相处之道。
针对上述问题,仍需加强野生动物保护生物学研究。在物种识别和种群调查方面,要应用人工智能、环境DNA等新技术方法,开展野生动物的智能物种识别、个体鉴定和快速物种多样性调查;在栖息地监测方面,要应用栖息地三维重建方法,识别野生动物适宜栖息地,为野化放归和廊道建设提供立体的栖息地选择方案;在遗传多样性评估方面,要应用人工智能、非损伤性遗传样本高通量测序方法,实现区域野生动物遗传多样性的快速评估。对于濒危物种解危技术和灭绝物种重现技术方面,要前瞻性保存濒危动物的细胞系等完整遗传资源,开展端粒到端粒的完整基因组测序,以及染色体人工合成、基因编辑、动物克隆等适用非模式野生动物的前沿性技术研发。对于保护生物学基础研究和保护实践脱节的问题,要引导科研人员开展保护需求导向的基础研究,更多地思考如何将基础研究成果转化为保护对策和保护实践工作中,从而真正提高野生动物保护成效。面对不断增多的人与濒危动物冲突的新问题,一方面要厘清冲突产生的原因、过程和危害,另外一方面要研发减缓冲突发生的防控技术以及完善损害赔偿机制,从而在濒危动物保护和人类生存需求之间寻找最佳的平衡。
参考文献
1 Hoffmann, M., Hilton-Taylor, C., Angulo, A., Böhm, M., Brooks, T. M., Butchart, S. H. M., Carpenter, K. E., Chanson, J., Collen, B., Cox, N. A., Darwall, W. R. T., Dulvy, N. K., Harrison, L. R., Katariya, V., Pollock, C. M., Quader, S., Richman, N. I., Rodrigues, A. S. L., Tognelli, M. F., Vié, J.-C., Aguiar, J. M., Allen, D. J., Allen, G. R., Amori, G., Ananjeva, N. B., Andreone, F., Andrew, P., Ortiz, A. L. A., Baillie, J. E. M., Baldi, R., Bell, B. D., Biju, S. D., Bird, J. P., Black-Decima, P., Blanc, J. J., Bolaños, F., Bolivar-G., W., Burfield, I. J., Burton, J. A., Capper, D. R., Castro, F., Catullo, G., Cavanagh, R. D., Channing, A., Chao, N. L., Chenery, A. M., Chiozza, F., Clausnitzer, V., Collar, N. J., Collett, L. C., Collette, B. B., Fernandez, C. F. C., Craig, M. T., Crosby, M. J., Cumberlidge, N., Cuttelod, A., Derocher, A. E., Diesmos, A. C., Donaldson, J. S., Duckworth, J. W., Dutson, G., Dutta, S. K., Emslie, R. H., Farjon, A., Fowler, S., Freyhof, J., Garshelis, D. L., Gerlach, J., Gower, D. J., Grant, T. D., Hammerson, G. A., Harris, R. B., Heaney, L. R., Hedges, S. B., Hero, J.-M., Hughes, B., Hussain, S. A., Icochea M., J., Inger, R. F., Ishii, N., Iskandar, D. T., Jenkins, R. K. B., Kaneko, Y., Kottelat, M., Kovacs, K. M., Kuzmin, S. L., La Marca, E., Lamoreux, J. F., Lau, M. W. N., Lavilla, E. O., Leus, K., Lewison, R. L., Lichtenstein, G., Livingstone, S. R., Lukoschek, V., Mallon, D. P., McGowan, P. J. K., McIvor, A., Moehlman, P. D., Molur, S., Alonso, A. M., Musick, J. A., Nowell, K., Nussbaum, R. A., Olech, W., Orlov, N. L., Papenfuss, T. J., Parra-Olea, G., Perrin, W. F., Polidoro, B. A., Pourkazemi, M., Racey, P. A., Ragle, J. S., Ram, M., Rathbun, G., Reynolds, R. P., Rhodin, A. G. J., Richards, S. J., Rodríguez, L. O., Ron, S. R., Rondinini, C., Rylands, A. B., Sadovy de Mitcheson, Y., Sanciangco, J. C., Sanders, K. L., Santos-Barrera, G., Schipper, J., Self-Sullivan, C., Shi, Y., Shoemaker, A., Short, F. T., Sillero-Zubiri, C., Silvano, D. L., Smith, K. G., Smith, A. T., Snoeks, J., Stattersfield, A. J., Symes, A. J., Taber, A. B., Talukdar, B. K., Temple, H. J., Timmins, R., Tobias, J. A., Tsytsulina, K., Tweddle, D., Ubeda, C., Valenti, S. V., Paul van Dijk, P., Veiga, L. M., Veloso, A., Wege, D. C., Wilkinson, M., Williamson, E. A., Xie, F., Young, B. E., Akçakaya, H. R., Bennun, L., Blackburn, T. M., Boitani, L., Dublin, H. T., da Fonseca, G. A. B., Gascon, C., Lacher, T. E., Mace, G. M., Mainka, S. A., McNeely, J. A., Mittermeier, R. A., Reid, G. M., Rodriguez, J. P., Rosenberg, A. A., Samways, M. J., Smart, J., Stein, B. A. & Stuart, S. N. 2010. The Impact of Conservation on the Status of the World’s Vertebrates. Science (New York, N.Y.)[J] 330, 1503-1509
2 Barnosky, A. D., Matzke, N., Tomiya, S., Wogan, G. O., Swartz, B., Quental, T. B., Marshall, C., McGuire, J. L., Lindsey, E. L. & Maguire, K. C. 2011. Has the Earth’s sixth mass extinction already arrived? Nature[J] 471, 51-57
3 Díaz, S. M., Settele, J., Brondízio, E., Ngo, H., Guèze, M., Agard, J., Arneth, A., Balvanera, P., Brauman, K. & Butchart, S. 2019. The global assessment report on biodiversity and ecosystem services: Summary for policy makers.
4 Zacharias, M. A. & Roff, J. C. 2001. Use of focal species in marine conservation and management: a review and critique. Aquatic conservation: marine and freshwater ecosystems[J] 11, 59-76
5 Zhao, S., Zheng, P., Dong, S., Zhan, X., Wu, Q., Guo, X., Hu, Y., He, W., Zhang, S. & Fan, W. 2013. Whole-genome sequencing of giant pandas provides insights into demographic history and local adaptation. Nature genetics[J] 45, 67-71
6 Zhan, X., Li, M., Zhang, Z., Goossens, B., Chen, Y., Wang, H., Bruford, M. W. & Wei, F. 2006. Molecular censusing doubles giant panda population estimate in a key nature reserve. Current biology[J] 16, R451-R452
7 胡锦矗. 1988. 大熊猫的繁殖生态学研究. 南充师院学报(自然科学版)[J], 79-83
8 Wei, F., Hu, Y., Yan, L., Nie, Y., Wu, Q. & Zhang, Z. 2014. Giant Pandas Are Not an Evolutionary cul-de-sac: Evidence from Multidisciplinary Research. Molecular biology and evolution[J] 32, 4-12
9 Zhang, B., Li, M., Zhang, Z., Goossens, B., Zhu, L., Zhang, S., Hu, J., Bruford, M. W. & Wei, F. 2007. Genetic viability and population history of the giant panda, putting an end to the “evolutionary dead end”? Molecular biology and evolution[J] 24, 1801-1810
10 Hu, Y., Wu, Q., Ma, S., Ma, T., Shan, L., Wang, X., Nie, Y., Ning, Z., Yan, L. & Xiu, Y. 2017. Comparative genomics reveals convergent evolution between the bamboo-eating giant and red pandas. Proceedings of the National Academy of Sciences[J] 114, 1081-1086
11 Nie, Y., Speakman, J. R., Wu, Q., Zhang, C., Hu, Y., Xia, M., Yan, L., Hambly, C., Wang, L. & Wei, W. 2015. Exceptionally low daily energy expenditure in the bamboo-eating giant panda. Science (New York, N.Y.)[J] 349, 171-174
12 Zhu, L., Wu, Q., Dai, J., Zhang, S. & Wei, F. 2011. Evidence of cellulose metabolism by the giant panda gut microbiome. Proceedings of the National Academy of Sciences[J] 108, 17714-17719
13 Huang, G., Wang, L., Li, J., Hou, R., Wang, M., Wang, Z., Qu, Q., Zhou, W., Nie, Y. & Hu, Y. 2022. Seasonal shift of the gut microbiome synchronizes host peripheral circadian rhythm for physiological adaptation to a low-fat diet in the giant panda. Cell Reports[J] 38
14 Zhu, L., Hu, Y., Qi, D., Wu, H., Zhan, X., Zhang, Z., Bruford, M. W., Wang, J., Yang, X. & Gu, X. 2013. Genetic consequences of historical anthropogenic and ecological events on giant pandas. Ecology[J] 94, 2346-2357
15 Hu, Y., Thapa, A., Fan, H., Ma, T., Wu, Q., Ma, S., Zhang, D., Wang, B., Li, M., Yan, L. & Wei, F. 2020. Genomic evidence for two phylogenetic species and long-term population bottlenecks in red pandas. Sci Adv[J] 6, eaax5751
16 Joshi, B. D., Dalui, S., Singh, S. K., Mukherjee, T., Chandra, K., Sharma, L. K. & Thakur, M. 2021. Siang river in Arunachal Pradesh splits red panda into two phylogenetic species. Mammalian Biology[J] 101, 121-124
17 Sun, X., Liu, Y.-C., Tiunov, M. P., Gimranov, D. O., Zhuang, Y., Han, Y., Driscoll, C. A., Pang, Y., Li, C. & Pan, Y. 2023. Ancient DNA reveals genetic admixture in China during tiger evolution. Nature ecology & evolution[J] 7, 1914-1929
18 Yang, L., Wei, F., Zhan, X., Fan, H., Zhao, P., Huang, G., Chang, J., Lei, Y. & Hu, Y. 2022. Evolutionary conservation genomics reveals recent speciation and local adaptation in threatened takins. Molecular biology and evolution[J] 39, msac111
19 Zhou, X., Guang, X., Sun, D., Xu, S., Li, M., Seim, I., Jie, W., Yang, L., Zhu, Q. & Xu, J. 2018. Population genomics of finless porpoises reveal an incipient cetacean species adapted to freshwater. Nature communications[J] 9, 1276